CRISPR(Clustered regularly interspaced short palindromic repeats)是生命进化历史上,细菌和病毒进行斗争产生的免疫武器,简单说就是病毒能把自己的基因整合到细菌,利用细菌的细胞工具为自己的基因复制服 务,细菌为了将病毒的外来入侵基因清除,进化出CRISPR系统,利用这个系统,细菌可以不动声色地把病毒基因从自己的染色体上切除,这是细菌特有的免疫 系统。微生物学家10年前就掌握了细菌拥有多种切除外来病毒基因的免疫功能,其中比较典型的模式是依靠一个复合物,该复合物能在一段RNA指导下,定向寻 找目标DNA序列,然后将该序列进行切除。许多细菌免疫复合物都相对复杂,其中科学家掌握了对一种蛋白Cas9的操作技术,并先后对多种目标细胞DNA进 行切除。这种技术被称为CRISPR/Cas9基因编辑系统,迅速成为生命科学最热门的技术。

这篇文章来自博德研究所(Broad Insititude)的大牛David Liu。这位在CRISPR领域颇有建树的年轻科学家,此次为我们带来了全新的进化版Cas酶——xCas9[1],比起目前使用最广泛的spCas9,xCas9在转录激活、DNA剪切、单碱基编辑等方面的应用范围扩大了四倍!这意味着,利用CRISPR-Cas9对目前已知致病突变修改的可能,从原来的不到三成猛涨到七成以上!与此同时,令研究者喜出望外的是,xCas9的脱靶效应比spCas9低得多得多,部分序列的试验数据仅有原始版的1/100!
这是我们认知范围里,能够应用在哺乳动物细胞中的、适用范围最广泛的Cas9变体,也是首次实现了适用范围、DNA特异性、剪切活性的全面提升。

刘大卫老师
要想说清楚xCas9为啥这么厉害,还得从Cas酶的限制说起。
大 家都知道,CRISPR/Cas系统原本是原核生物为了应付外源遗传物质入侵的一种防卫机制,所谓的CRISPR序列,就是敌人的“黑名单”。每当有一个 新的敌人入侵,CRISPR/Cas系统就会剪下它的一段基因作为“身份证”,插入CRISPR序列中保存起来,以备下一次敌袭。我们科学家也正是利用 CRISPR/Cas系统的这个特点来实现对目标基因的剪切和替换。
但是啊,这段剪下来的“身份证”可不是随意选择的,它们的附近往往具有一段特殊的序列,我们把它叫做原间隔序列临近基序(PAM)。这段序列根据Cas酶的种类不同而有所不同,像现在科学家最常使用、最成熟的spCas9,主要识别的就是NGG序列(N指任意碱基)。spCas9也是目前PAM适用最广泛的Cas酶。
这个最广泛有多广泛?我们简单计算一下,DNA存在四种碱基,NGG出现的可能性只有十六分之一! 对于沉默特定基因来说,这个范围差不多够用了,但是近年来,单碱基编辑技术不断走向成熟,这项技术要求PAM位于目标碱基的15±2个核苷酸范围内 [2,3],spCas9显然不能满足研究者的需求。而且如果能够进一步把PAM适应范围扩大,让其落在DNA同源重组位点附近,重组效率也能够获得一定 的提升[4,5]。
想要改变Cas酶的功能就要改变蛋白的结构。传统实验室方法是在蛋白的编码序列中人为制造突变,再进行功能筛查。但是想一想,这效率也太低了,一个个合成再筛选得筛到哪年去,累死科学家啊。
David Liu不怕,他有个独门杀器,噬菌体辅助持续进化器(PACE)[6]!简单来说,就是把需要突变的蛋白基因序列放进噬菌体基因组,利用噬菌体生命周期短暂的特点,进行大批量持续性的突变。噬菌体所寄宿的大肠杆菌内含有诱导突变的质粒,能够加速突变的发生。噬菌体最短十分种就能传代一次,一周内就能够实现成百上千次的传代变异,还完全不用科学家插手,比传统方法效率提高了100倍。
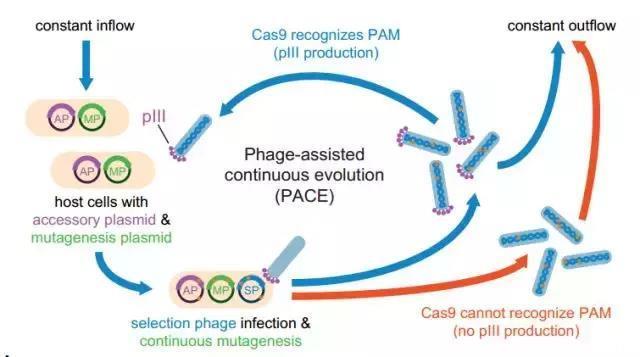
在编码的时候,把Cas酶编码成噬菌体存活的必需基因,这样就可以利用其生命周期自动筛选掉没有表达的噬菌体了
利用这个独门杀器,研究者们很快筛选出了一批PAM适用更广泛的Cas酶——xCas9,其中xCas9(3.7)效果最好,xCas9(3.6)次之。
研究者决定对xCas9的真本事在人细胞中做一下测试。在激活转录这一点上,可以说xCas9(3.7)简直完胜,针对各PAM效率比spCas9高出数倍不止;切割DNA的能力,xCas9也完全没在怕的,spCas9的老强项NGG上已经赢过一头,对spCas9无能为力的NG、GAA、GAT上均表现出了不俗的能力。这意味着,如果使用xCas9,CRISPR/Cas系统的适用范围至少扩大了四倍!
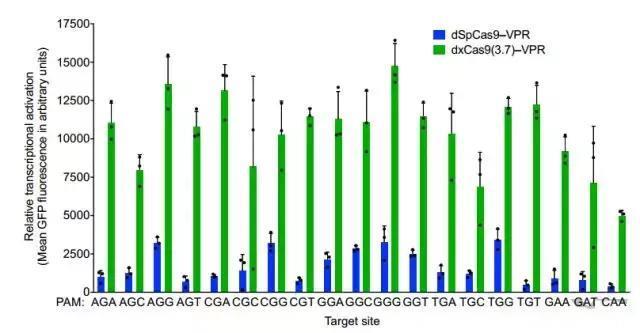
转录激活上xCas9(绿)完胜
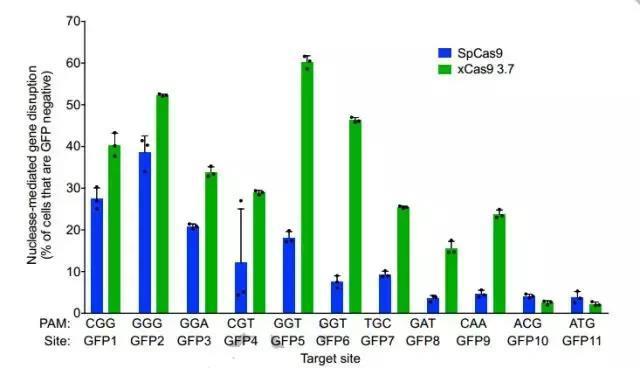
DNA切割能力上xCas9也明显更胜一筹
这个扩大的PAM范围拿到单碱基编辑上,则更令科学家们振奋了,毕竟目前为止,单碱基编辑器还只限于NGG这一段PAM,能够进行修改的序列很有限。xCas9到手,单碱基编辑的适用范围也大大增加。
根据ClinVar数据库[9]显示,能够利用C•G→T•A修正的致病突变共有4422个,原本单碱基编辑的适用范围只有26%,xCas9使这个数字跃升到73%!同样,能够利用A•T→G•C修正的致病突变有14969个,修正可能从28%跃升到71%!
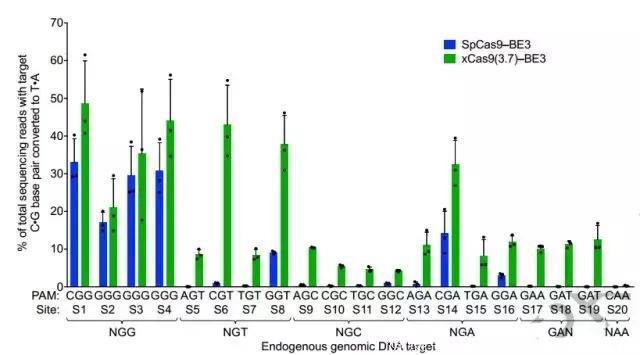
单碱基编辑的适用范围也大大增加
PAM适用范围扩大了这么多,CRISPR原本就是个问题的脱靶效应想必更严重了吧。。。然而并没有!研究者在对常见的几条基因序列进行了模拟,发现xCas9(3.7)的脱靶效应竟然远低于spCas9!在EMX1序列中,spCas9的脱靶效应接近百分之二十,xCas9(3.7)居然百分之百“命中红心”;HEK site4和VEGFA这样有名的复杂基因,xCas9(3.7)命中率也有4-9倍的提升,真是厉害了!
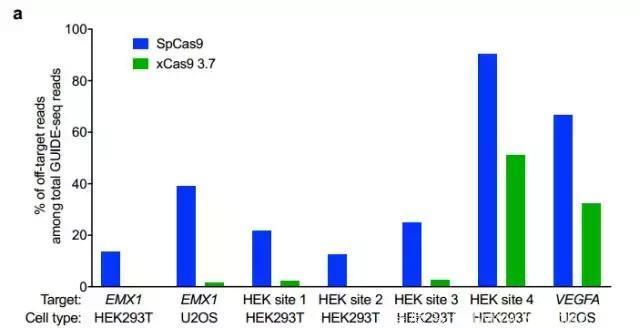
xCas9的脱靶效应超乎意料的低

100%!!!
遗憾的是,xCas9为什么实现更广泛PAM 的同时,脱靶效应还低了,这个问题并没有得到解决,David Liu面对采访坦然回答“不知道”。
Liu在采访中指出,spCas9经过多年的实验验证,但xCas9只经过几十个位点的测试,并不能百分百肯定xCas9就比spCas9更好(他谦虚了)。但不可否认的是,这种双赢的局面十分惊人,已经有科学家表示想要赶快在自己的实验室中使用xCas9。
话说PACE这项技术也实在是很厉害,赶快拿来改造下其他的Cas酶,估计还有大发现!
参考资料:
[1] https://www.nature.com/articles/nature26155
[2] Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A. & Liu, D. R. Programm editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420–424, doi:10.1038/nature17946 (2016)
[3] Gaudelli, N. M. et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature 551, 464–471, doi:10.1038/nature24644(2017)
[4] Findlay, G. M., Boyle, E. A., Hause, R. J., Klein, J. C. & Shendure, J. Saturation editing of genomic regions by multiplex homology-directed repair. Nature 513,120–123, doi:10.1038/nature13695 (2014)
[5] Yang, L. et al. Optimization of scarless human stem cell genome editing.Nucleic Acids Res 41, 9049–9061, doi:10.1093/nar/gkt555 (2013)
[6] https://www.nature.com/articles/nature09929
[7]http://www.sciencemag.org/news/2018/02/upgrade-makes-genome-editor-crispr-more-muscular-precise
[8] https://www.nature.com/articles/d41586-018-02540-x
[9]Landrum, M. J. et al. ClinVar: public archive of relationships among sequence
variation and human phenotype. Nucleic Acids Res 42, D980–985,
doi:10.1093/nar/gkt1113 (2014).